Giugno 2004 - Volume VII - numero 6
M&B Pagine Elettroniche
Pediatria per l'ospedale
L'epatite
B
(parte
prima)
Ormai le
due coorti di vaccinati, quella dei nuovi nati e quella dei dodicenni
si sono incontrate: abbiamo in Italia una copertura vaccinale del 95%
contro l'epatite B per 24 classi di età. D'ora innanzi
vaccineremo solo i nuovi nati. Il successo ha portato alla quasi
scomparsa dell'infezione da virus dell'epatite B nella parte
della popolazione sottoposta alla vaccinazione. Ma con due milioni
circa di portatori cronici dell'HBV l'herd immunity non ha potuto
esplicare appieno la sua funzione di riduzione della circolazione
dell'HBV.
E'
venuto il momento di rivisitare la storia naturale della malattia e
le sue conseguenze cliniche: un articolo di revisione (Ganem
D, Prince AM – Hepatitis B virus infection – Natural hystory and
clinical conseguences – N Engl J Med 2004, 350:1118-29) ci
aiuterà in questo percorso.
Il virus
dell'epatite B (HBV) è il membro prototipo della famiglia
delle Hepadnaviridiae (hepatotropic DNA virus). Questi virus hanno la
caratteristica d'invadere le cellule del fegato, anche se piccole
quantità di questo virus si ritrovano nel rene, nel pancreas e
nelle cellule mononucleate. Tuttavia l'infezione in queste sedi
extraepatiche non si accompagna costantemente alla comparsa di quadri
clinici.
I virioni
dell'HBV hanno un diametro di 40-42 nm con un rivestimento esterno
di lipoproteine, che contiene tre rivestimenti di glicoproteine (o
antigeni di superficie = HBsAg). All'interno del rivestimento si
trova il nucleocapside virale o core. Il core
contiene il genoma virale, un DNA circolare, parzialmente doppio, di
3,2 kb e una polimerasi che è responsabile della sintesi del
DNA virale nelle cellule infettate. Lo studio del DNA dei molti ceppi
isolati di HBV ha confermato l'esistenza di molti genotipi virali,
ognuno dei quali ha una distribuzione geografica caratteristica, ma
non si accompagna a caratteristiche cliniche specifiche.
Oltre al
virione, le cellule infettate con l'HBV producono due distinte
particelle lipoproteiche:
- le sfere di 20 nm di diametro
- le forme filamentose di diametro simile ma di lunghezza diversa.
Queste
particelle di HBsAg contengono solo le glicoproteine di rivestimento
e i lipidi derivati dalla parete delle cellule dell'ospite e sono
in numero molto superiore ai virioni: le sfere in una proporzione di
10.000 a 1 e le forme filamentose in una proporzione di 1.000 a 1.
Geni
e proteine virali
Il genoma
dell'HBV ha soltanto 4 lunghe aperte strutture di lettura.
Laregione preS-S (presuperficie-superficie) codifica 3
antigeni virali di superficie con differenti inizi di traduzione per
ognuno dei tre. La proteina più abbondante è la
proteina 24-kD S chiamata HBsAg. L'inizio del codone a monte genera
la proteina M (o preS2), la cui funzione è sconosciuta.
L'inizio del codone ancora più a monte codifica per la
proteina L (o pre-S1), che si pensa giochi un ruolo nel legame del
virus alla cellula ospite e nell'assemblaggio del virione e nel suo
rilasciamento dalle cellule.
Gamen
D, Prince AM – Hepatitis B virus infection –Natural history and
clinical consequences – N Engl J Med 2004, 350:1118-29
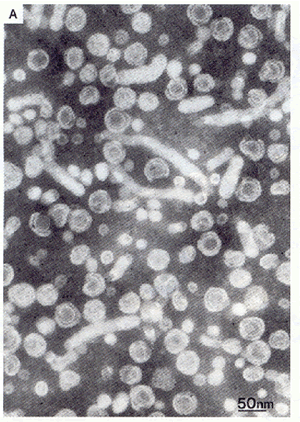
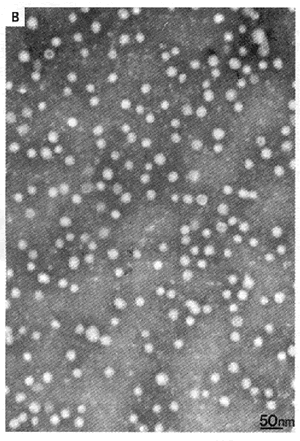
Figura
1 - Struttura delle particelle HBsAg associate
Nella
parte A si osservano i virioni del virus dell'epatite B (particella
Dane) e filamenti
Nella
parte B si osservano le particelle di HBsAg da 20 nm
L'antigene
HBs induce la sintesi di anticorpi, che hanno un valore protettivo,
attiva le cellule della memoria sia B che T e inoltre induce risposte
di tipo cellulare.
La
regione preC-C (precore core) codifica per l'antigene del core
dell'HBV (ABcAg) e per l'antigene e (HBeAg). Quando le catene
attraversano il complesso di Golgi, la scissione da parte delle
proteasi cellulari genera l'HBeAg, un frammento di 16-kD che viene
secreto nel sangue. L'HBeAg non gioca un ruolo nell'assemblaggio
del virus e la sua funzione non è chiara.
La
regione P codificante è specifica della polimerasi virale, un
enzima multifunzionale interessato nella sintesi del DNA e nella
incapsidazione RNA. La struttura aperta di lettura (open reading
frame) X codifica per la proteina virale X (HBx) che modula il
segnale di transduzione della cellula ospite e può
direttamente o indirettamente interessare l'ospite e l'espressione
del gene virale. L'attività della proteina X è
assolutamente richiesta per la moltiplicazione in vivo e per la
diffusione del virus.
Ciclo
di moltiplicazione virale
La figura
2 mostra i principali aspetti del ciclo moltiplicativo degli
epadnavirus, l'aspetto principale dei quali è la
moltiplicazione del genoma DNA da parte della trascrizione inversa di
un intermediario RNA.
Per prima
cosa i virioni si legano ai recettori della superficie cellulare, non
ancora identificati. Dopo la fusione della membrana, il core
attraversa il citosol (cioè la parte liquida del citoplasma:
il citosol corrisponde cioè al citoplasma meno gli organelli e
altri componenti insolubili in esso contenuti) e giunge al nucleo,
dove il suo genoma DNA viene convertito in una forma circolare
chiusa, covalente, che serve come templato di trascrizione per la RNA
polimerasi II dell'ospite. Questo enzima genera una serie di
costituenti genomici e subgenomici.
Tutti gli
RNA virali sono trasportati nel citoplasma, dove vengono tradotti nel
rivestimento, nel core, nelle proteine della polimerasi e anche nei
polipeptidi X e preC. Successivamente i nucleocapsidi sono assemblati
nel citosol e durante questo processo una molecola di RNA genomico è
incorporata nel core virale.

Figura
2 – La moltiplicazione dell'HBV
Il
virione HBV si lega ai recettori sulla superficie della cellula e
penetra nel suo interno. La particella core dell'HBV migra nel
nucleo dell'epatocita, dove il suo genoma viene modificato in un
DNA circolare chiuso covalente (cccDNA) che rappresenta il templato
per la trascrizione dell'RNA messaggero virale (mRNA). L'mRNA
virale che ne risulta viene trasferito nel citoplasma per produrre la
superficie virale, il core, la polimerasi e la proteina X. Quindi la
progenie del capside virale si assembla, incorporando l'RNA virale
del genoma. Questo RNA è trascritto inversamente a DNA. Il
core che si viene a formare può sia passare nel reticolo
endoplasmico per essere avvolto e per uscire dalla cellula, oppure il
suo genoma può essere riciclato nel nucleo per la conversione
a ccDNA. Le piccole sfere, di colore pesca, all'interno del core
sono la DNA polimerasi virale.
A questo
punto l'RNA virale viene incapsulato e inizia la trascrizione
inversa. La sintesi delle due eliche di DNA è sequenziale.
Come si
vede dalla figura, alcuni core sono trasportati di nuovo nel nucleo,
per subire la elaborazione già descritta in precedenza, mentre
la maggior parte di essi raggiunge la regione delle membrane
intracellulari per formare le proteine del rivestimento virale. E'
a questo punto che acquisiscono i rivestimenti lipoproteici, che
contengono gli antigeni di superficie L, M ed S; in seguito il virus
lascia la cellula.
Patogenesi
dell'epatite B
Fin
dall'inizio degli studi sull'epatite B, apparve chiaro che il
ciclo di moltiplicazione del virus nella cellula non è
direttamente citotossico.
Questo
fatto è in accordo con l'osservazione che la maggior parte
dei portatori di HBV è asintomatica e ha minime lesioni del
fegato, nonostante che la moltiplicazione virale intraepatica sia
intensa e persistente. Si pensa che siano le risposte immuni
dell'ospite verso gli antigeni virali, presenti negli epatociti
infettati, i principali determinanti delle lesioni epatiche. Questa
affermazione è in accordo con l'osservazione clinica che
pazienti con deficit immunologici, infettati con HBV, hanno spesso
lesioni acute lievi del fegato, ma hanno alte probabilità di
essere portatori cronici.

Figura
3- Risposte immuni cellulari all'HBV
L'HBV
si moltiplica nell'epatocita per produrre particelle HBsAg e
virioni. Ambedue i tipi di particelle sono catturati dalle cellula
presentanti l'antigene, che degradano le proteine virali a peptidi
che sono presenti sulla superficie cellulare, legati alle molecole
MHC di classe I e II. Questi antigeni peptidici possono essere
riconosciuti dalle cellule T CD8 positive o CD4 positive.,
rispettivamente, che sono in tal modo sensibilizzate. Le cellule T
citotossiche virus specifiche CD8 positive (con l'aiuto delle
cellule T CD4 positive) possono riconoscere gli antigeni virali
presenti sulle catene MHC classe I presenti sugli epatociti
infettati. Questa reazione di riconoscimento può portare sia a
una diretta lisi dell'epatocita infetto, sia alla liberazione di
interferon gamma e di tumor necrosis factor alfa, che possono
regolare verso il basso la moltiplicazione virale negli epatociti
circostanti, senza uccidere direttamente la cellula.
Le
risposte immuni all'HBV e il loro ruolo nella patogenesi
dell'epatite B non sono completamente conosciute. Studi clinici
correlati mostrano che sono evidenti nel sangue periferico di
soggetti con epatite B acuta autolimitantesi, forti risposte delle
cellule T verso i principali antigeni dell'HBV. Le risposte
antivirali del linfociti T citotossici sono dirette verso epitopi
multipli all'interno del core, della polimerasi e delle proteine
del rivestimento dell'HBV; sono state dimostrate anche forti
risposte delle cellule T helper verso le proteine C e P. Al contrario
nei portatori cronici di HBV, tali risposte virus-specifiche delle
cellule T sono fortemente attenuate, come risulta dall'esame delle
cellule del sangue periferico. Anche le risposte anticorpali sono
forti e durature in ambedue le situazioni (sebbene gli anticorpi
verso l'HBsAg (anti-HBs) non siano dimostrabili nei portatori per
l'eccesso di HBsAg circolante). Questo quadro suggerisce fortemente
che le risposte delle cellule T, specialmente del linfociti T
citotossici, giocano un ruolo centrale nella clearance virale,
Nella
figura 3 sono riportati i tipi principali della risposta cellulare
immune.
I
meccanismi con i quali i linfociti T citotossici uccidono le cellule
epatiche e la causa della clearance virale sono stati a lungo
studiati nel topo transgenico. Poiché questi topi albergano i
geni dell'HBV nel loro DNA, essi sono largamente tolleranti alle
proteine dell'HBV e quindi, secondo quanto sopra esposto, non
sviluppano lesioni epatiche clinicamente significative (Chisari FV,
Curr Top Microbiol Immunol 1996, 206:149-73). Tuttavia se linfociti T
citotossici antivirali di animali singenici sono trasferiti in questi
topi, si sviluppano lesioni epatiche acute con la maggioranza degli
aspetti dell'epatite B clinica. E' importante rilevare che in
questo modello , il numero di epatociti uccisi è molto piccolo
e insufficiente a determinare un danno del fegato. Questo rilievo
suggerisce che la maggior parte delle lesioni è dovuta alle
risposte infiammatorie non specifiche, messe in moto dalla risposta
dei linfociti T citotossici, come il tumor necrosis factor, i
radicali liberi e le proteasi. Altre popolazioni delle cellule
immuni, principalmente le cellule natural killer probabilmente
contribuiscono alle lesioni del fegato.
Recenti
esperimenti suggeriscono che alcuni prodotti dell'infiammazione,
principalmente l'interferon gamma e il tumor necrosis factor alfa
possono avere un effetto antivirale che non interessa l'uccisione
delle cellule bersaglio. Infatti quando linfociti T citotossici sono
trasferiti al topo, che presenta HBV in moltiplicazione, il DNA e
l'RNA virale scompaiono rapidamente dal fegato: un effetto che può
essere bloccato dalla somministrazione di anticorpi diretto verso le
due citochine
Vuoi citare questo contributo?